Python連携機能を用いた全エネルギーの体積依存性の予測#
Advance/NanoLaboではPythonスクリプトを介してさまざまな操作を行うことが可能です。Pythonスクリプトを活用することで煩雑なワークフローを自動化することができます。
本事例ではゲルマニウム単結晶の全エネルギーがユニットセルの体積にどのように依存するかを解析します。体積が異なる31個のモデルについて、Pythonスクリプトを用いて、モデルの作成および計算結果の解析を行います。
Warning
本ページではAdvance/NanoLaboの機能紹介を目的としてサンプルコード1を記載していますが、その動作の保証はしかねます。Python連携機能の使用方法はオンラインマニュアルをご覧ください。
体積が異なるモデルの作成#
ゲルマニウム単結晶の最適化構造2をPythonスクリプトから読み込み、新しいプロジェクトを作成します。新たに作成したプロジェクトについて、ユニットセル3を等方的に拡大(縮小)します。拡大(縮小)率を変えながらこの手続きを繰り返すことで、体積の異なる複数個のプロジェクトを作成することができます。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | |
計算実行#
プロジェクト名を指定してrun_project関数を呼び出すことで、計算を実行します。
1 2 3 | |
体積依存性の解析#
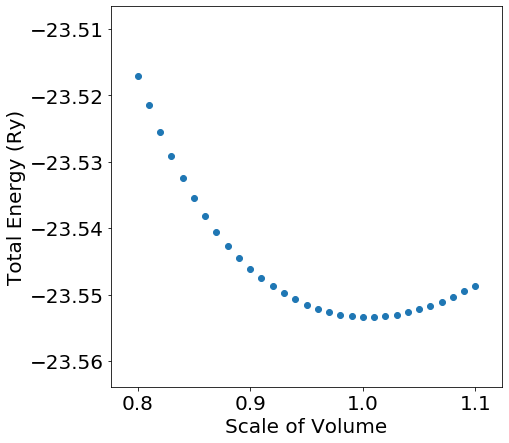
計算終了後にqe_get_total_energy関数を呼び出すことで、系の全エネルギーを取得することができます。本事例では体積の異なる31個のプロジェクトから全エネルギーを読み出し、体積比―全エネルギープロットを描画しています。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 | |
平衡構造と比較して、体積比が以内の領域では、全エネルギーの変化はに満たないですが、ほど圧縮されるとだけ不安定化することが分かります。
このような全エネルギーの体積依存性の解析を行うことで、体積弾性率や転移圧力を予測することも可能になります。
関連ページ#
-
サンプルコードはPython3.7以上を前提としています。サンプルコード中で NumPyとMatplotlibを使用しています。これらについては各ライブラリの公式ウェブサイトをご覧ください。 ↩
-
構造最適化計算をAdvance/NanoLabo上で実行し、その結果をcifファイル(Ge-Optimized.cif)にあらかじめ保存しています。 ↩
-
ユニットセルだけでなく原子座標の編集もPythonから行うことが可能です。 ↩